Control de Calidad Metagenomas
FASTQC
Los secuenciadores de alto rendimiento pueden generar cientos de millones de secuencias en una sola ejecución. Antes de sacar conclusiones biológicas, siempre debe realizar comprobaciones de control de calidad para asegurarse de que los datos sin procesar se vean bien y que no haya problemas o sesgos en los datos.
FastQC tiene como objetivo proporcionar un informe de control de calidad que puede detectar problemas que se originan en el secuenciador o en el material de la biblioteca inicial.
El comando para verificar nuestros datos es:
| fastqc metagenoma_1.fastq metagenoma_2.fastq -o quality_bbduck/ |
BBDUCK
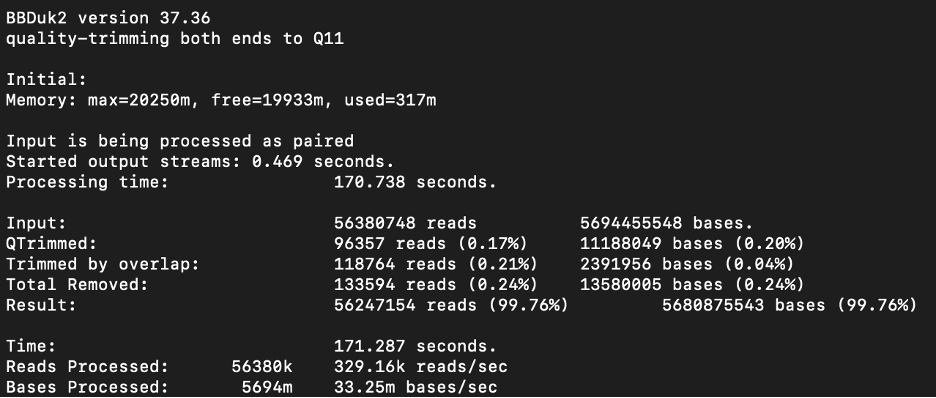
BBDuk es capaz de recortar y filtrar calidad, recortar adaptadores, filtrar contaminantes mediante coincidencia k-mer, enmascaramiento de secuencias, filtrado GC, generación de histograma, submuestreo entre otros. Específicamente, cualquier combinación de operaciones es posible en una sola corrida, con la excepción de las operaciones basadas en k-mer (recorte de k-mer, enmascaramiento de k-mer o filtrado de k-mer); a lo sumo 1 operación basada en k-mer se puede hacer en una sola pasada. BBDuk2 permite múltiples operaciones basadas en k-mer en una sola pasada, y es equivalente a BBDuk.
|
bbduk2.sh in=Mix1.fastq in2=Mix2.fastq out=Mix1-bbduk.fastq out2=Mix2-bbduk.fastq minlen=100 qtrim=rl trimq=11 tpe tbo |
FASTQC
Ahora se puede volver a ejecutar la herramienta FastQC para realizar comprobaciones de control de calidad para asegurarse de que los datos se vean bien y que no haya problemas con los datos.
| fastqc bbduck-mix_1.fastq bbduck-mix_2.fastq -o quality_bbduck/ |


