Análisis de diversidad beta
También se puede medir la diversidad beta en Qiita. Las medidas de diversidad beta presentan la rotación entre muestras (es decir, la diversidad entre muestras en lugar de dentro de cada muestra). Esto se utiliza para comparar muestras entre sí.
Disimilitud de Bray-Curtis
Una métrica de diversidad beta comúnmente utilizada es la disimilitud de Bray-Curtis. Esta métrica cuantifica que tan diferentes son las muestras entre sí.
Para realizar un análisis de la diversidad beta utilizando la métrica de disimilitud de Bray-Curtis, seleccione el artefacto "Rarefy table(BIOM)" en la red de procesamiento y seleccione "Process". Luego, seleccione “Beta diversity” en el menú desplegable. Los parámetros aparecerán debajo del diagrama de flujo de trabajo:
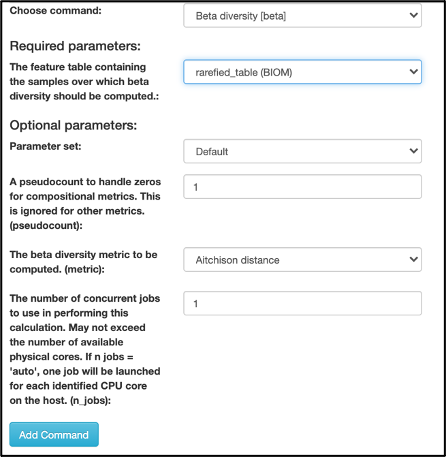
Se han seleccionado automáticamente varios parámetros. En el campo, “The beta diversity metric… (metric)”, se especifica el análisis de diversidad beta que se ejecutará. Seleccione " Bray-Curtis dissimilarity" en el menú desplegable de este cuadro y haga clic en "Add Command". Una vez ejecutado el flujo de trabajo, se desplegará el siguiente resultado:
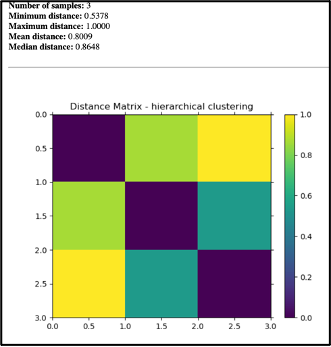
Para crear una gráfica de coordenadas principales de la matriz de distancias de disimilitud de Bray-Curtis, seleccione el artefacto “distance matrix (distance matrix)” y seleccione “Process”. Seleccione “Perform Principal Coordinate Analysis (PCoA)” en el menú desplegable. Los parámetros aparecerán debajo del diagrama de flujo de trabajo y seleccionados de manera predeterminada:
Al hacer clic en el artefacto “pcoa (ordination_results)” (Análisis de coordenadas principales) se abrirá una visualización interactiva de la similitud entre sus muestras. En términos generales, cuanto más similares sean las muestras con respecto a sus características, es probable que estén más cerca de la gráfica de ordenación de PCoA. El programa de visualización Emperor ofrece una forma muy útil de explorar cómo los patrones de similitud en sus datos se asocian con diferentes categorías de metadatos.
Una vez que se carga el programa de visualización Emperor, el resultado de PCoA se verá así:
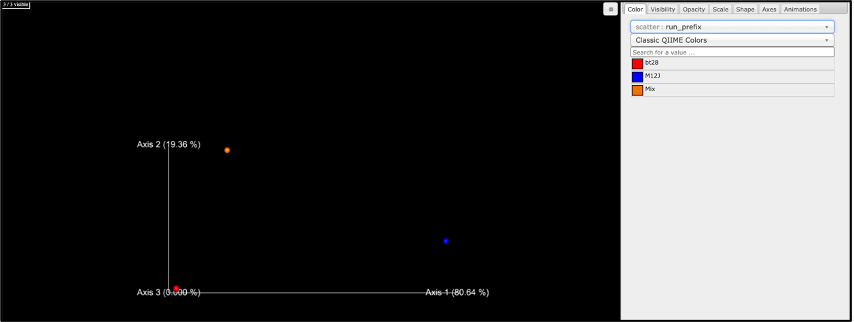
Análisis UniFrac no ponderado
Otra métrica de distancia comúnmente utilizada para medir la diversidad beta es la distancia UniFrac no ponderada. No ponderado se refiere a que la métrica considera solo la riqueza de características y no la abundancia, al comparar muestras entre sí. Esto difiere de la métrica de distancia ponderada de UniFrac, que tiene en cuenta tanto la riqueza como la abundancia de características para cada muestra.
Para realizar un análisis UniFrac no ponderado, seleccione el artefacto "rarefied table (BIOM)" en la red de procesamiento y seleccione "Process". Luego, seleccione " Beta diversity (phylogenetic)" en el menú desplegable. Los parámetros aparecerán debajo del diagrama de flujo de trabajo:
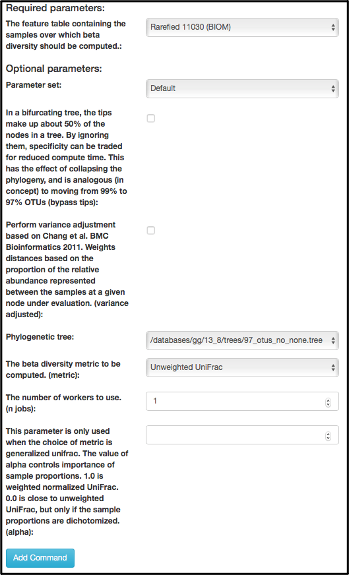
Para crear una gráfica de coordenadas principales de la matriz de distancia Unifrac no ponderada, seleccione el artefacto "distance_matrix (distance_matrix)" que se genera utilizando la distancia UniFrac no ponderada. Una vez que haya seleccionado el artefacto de la matriz de distancia, seleccione "Perform Principal Coordinate Analysis (PCoA)" en el menú desplegable. Los parámetros aparecerán debajo del diagrama de flujo de trabajo
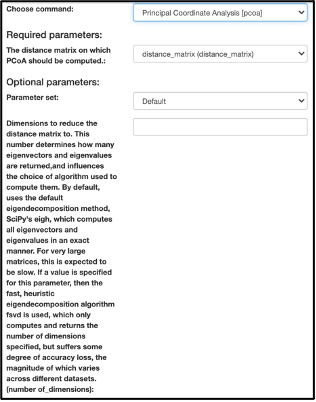
Una vez realizado el análisis se abrirá una visualización interactiva de la similitud entre sus muestras. En términos generales, cuanto más similares sean las muestras con respecto a sus características, es probable que estén más cerca de la gráfica de ordenación de PCoA. El programa de visualización Emperor ofrece una forma muy útil de explorar cómo los patrones de similitud en sus datos se asocian con diferentes categorías de metadatos.

Significancia del grupo de diversidad beta
Otra forma de estudiar la diversidad beta es midiendo la importancia del grupo de diversidad beta. La significancia del grupo de diversidad beta mide si los grupos de muestras son significativamente diferentes entre sí mediante una prueba estadística basada en permutación. Los grupos de muestra se designan mediante variables de metadatos.
Si ha completado el tutorial hasta este punto, puede comenzar el análisis de la importancia del grupo de diversidad beta desde una de las matrices de distancia de diversidad beta ya creadas.De lo contrario, para realizar un análisis de significancia de grupo beta, seleccione el artefacto de "rarefied table (BIOM)" en la red de procesamiento y seleccione "Process". Seleccione "Beta Diversity" en el menú desplegable. Los parámetros aparecerán debajo del diagrama de flujo de trabajo de manera predeterminada.
Para crear el análisis de significancia del grupo beta, seleccione el artefacto “distance_matrix (distance_matrix)” de interés en la red de Procesamiento y seleccione “Beta diversity group significance” en el menú desplegable. Los parámetros aparecerán debajo del diagrama de flujo de trabajo, en el campo “Metadata column to use” que se usará para determinar la importancia entre grupos en este caso utilizamos “Runid”. Usando la casilla de verificación "Perform pairwise tests…" podemos indicar si queremos que la significación del grupo se ejecute "Pairwise", de lo contrario, el análisis se realizará en todos los grupos (Non-pairwise):
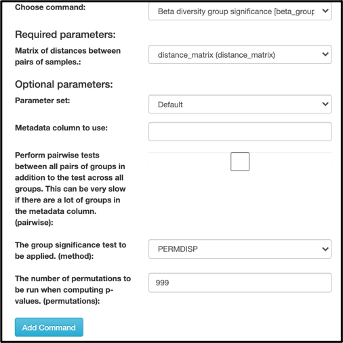
Una vez que se elige el artefacto de “visualization (q2_visualization)” de significación del grupo beta en la red, aparecerá la descripción general de significación del grupo de diversidad beta, que en nuestro caso muestra los resultados de PERMANOVA (es decir, en todos los grupos) y las gráficas de significación del grupo:
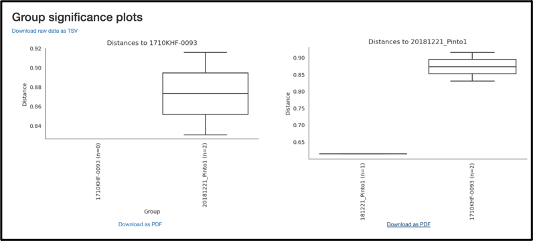

Los resultados de las pruebas PERMANOVA por pares también se mostrarán si se incluyen en el análisis:
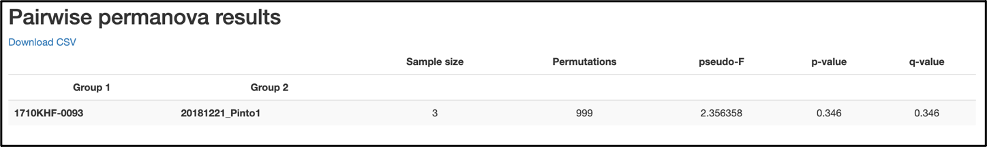
El comando “Beta diversity group significance” proporciona PERMANOVA que se puede ejecutar en una única variable de metadatos categórica.